MetaPhlAn3.0が利用できるようですね。MetaPhlAn3なのか3.0なのか・・・。公式には3.0なので3.0でしょう。変更点として下記が挙げられています。
- 新しいクレードマーカー遺伝子(New MetaPhlAn marker genes extracted with a newer version of ChocoPhlAn based on UniRef)
- 不詳な微生物のメタゲノムも予測可能(Estimation of metagenome composed by unknown microbes with parameter --unknown_estimation)
- -index latestを指定することで最新のデータベースを随時ダウンロード可能(Automatic retrieval and installation of the latest MetaPhlAn database with parameter --index latest)
- VIrus profilingが可能(2.0でも出来なかったっけ・・・)(Virus profiling with --add_viruses)
- メタゲノムのサイズ計算によるクレードにマップされたリードの予測(精度)改善(Calculation of metagenome size for improved estimation of reads mapped to a given clade)
- NCBI taxIDがアウトプットに追加された(Inclusion of NCBI taxonomy ID in the ouput file)
- CAMI形式のアウトプットを選択可能(CAMI (Taxonomic) Profiling Output Format included)
- MAPQ値の低いリードの除去(Removal of reads with low MAPQ values)
github.com
huttenhower.sph.harvard.edu
HUMAnN3はまだofficialには公開されていないようです。
humann3 – The Huttenhower Lab
add HUMAnN3 to MetaPhlAn3 Docker container · Issue #6 · waldronlab/curatedMetagenomicDataHighLoad · GitHub
biocondaでのインストールが失敗したのでpipより。
git clone -b 3.0 https://github.com/biobakery/MetaPhlAn.git pip install metaphlan metaphlan --help
長いサンプルやヘルプ表示が出たので割愛。
重要そうなのは新しいfeatureでしょうか。
- -x INDEX, --index INDEX
Specify the id of the database version to use. If "latest", MetaPhlAn will get the latest version. If the database files are not found on the local MetaPhlAn installation they will be automatically downloaded [default latest]
- --min_mapq_val MIN_MAPQ_VAL
Minimum mapping quality value (MAPQ)
- --CAMI_format_output Report the profiling using the CAMI output format
- --unknown_estimation Ignore estimation of reads mapping to unkwnown clades
- --add_viruses Allow the profiling of viral organisms
今回はヒトゲノムのコンタミを除去したPaired-end readsをinputとして試行してみます。
サンプルはhttps://www.ncbi.nlm.nih.gov/sra/?term=SRR9040524です。
あえてoralで、新しいfeatureを利用してprofiling。
metaphlan notal.SRR9040524.1.fastq,notal.SRR9040524.2.fastq --input_type fastq -x latest --bt2_ps very-sensitive --force --bowtie2out SRR9040524.bowtie2.bz2 --nproc 12 -t rel_ab -o SRR9040524_metaphlan3_output.txt --legacy-output --unknown_estimation --add_viruses
最新データベースのダウンロード、Bowtie2 DBの作成が自動的に始まりました。
(mpa_v296_CHOCOPhlAn_201901_marker_info.txt.bz2)
MetaPhlAn2の結果と比較(のために--unknown_estimation, --add_virusesはoff)。
metaphlan notal.SRR9040524.1.fastq,notal.SRR9040524.2.fastq --input_type fastq -x latest --bt2_ps very-sensitive --force --bowtie2out SRR9040524.bowtie2.bz2 --nproc 12 -t rel_ab -o SRR9040524_metaphlan3_output.txt --legacy-output
同定されたtaxonの数
| version | phylum | class | order | family | genus | species |
| 2 | 4 | 6 | 9 | 10 | 11 | 26 |
| 3 | 4 | 6 | 8 | 9 | 9 | 15 |
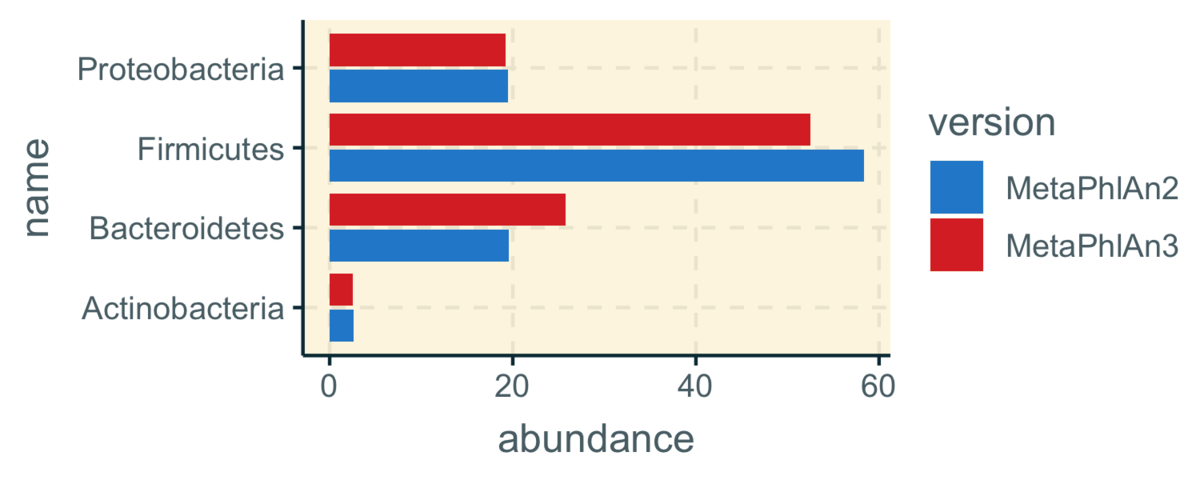
Phylumのabundance(百分率, ggthemr)
Speciesのabundance(百分率, ggthemr)
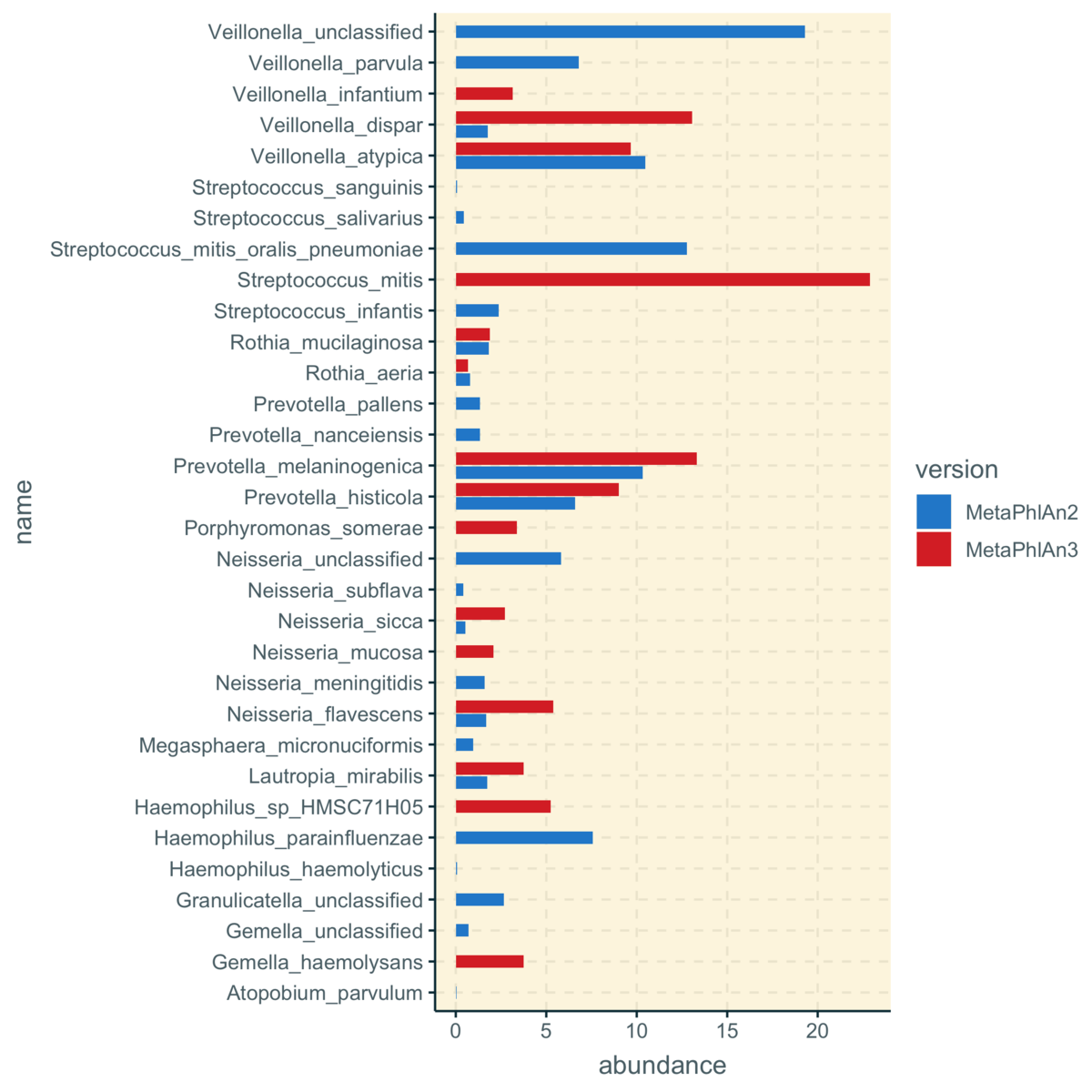
全体にMetaPhlAn3.0の方が同定されるtaxonが少ない印象。Firmicutesは結構違いますね(3.0: 52.5% vs 2: 58.3%)。Speciesレベルも・・・今までのMetaPhlAn使った報告はどう解釈すればいいのやら・・・。
どちらが正確かなどはしっかりベンチマークしないとですが、MetaPhlAn2についてはレビュー論文やTwitterなどでもメモリ使用量は抑えられるが精度がちょっと・・・というような評価があったので期待。
アルゴリズム的にあまり信用していないMetaPhlAn2、と言っても彼らの十八番のヒト腸内メタなら大丈夫だろうと思って種組成解析目的で使ってみたが、確認用にやった種名のマニュアルアノテーションの結果とClostridia等が全然一致しなくてヤバさを実感。詳細な種組成解析をやれるツールではないなこれ。
— Tyu_Shi (@Tyu_Shi) February 10, 2020